SBX: Sequenzierung neu gedacht
Die Schweizer Roche hat eine völlig neuartige Sequenzierungstechnologie vorgestellt. Mit der skalierbaren und flexiblen „Sequenzierung durch Expansion“ (SBX) lässt sich die Genomsequenzierung enorm beschleunigen.
Im Gegensatz zu herkömmlichen Techniken wie der „Sequenzierung durch Synthese“ (SBS), wie sie auch der Branchenführer Illumina einsetzt, beruht Roches neues chemisches Verfahren auf der Expansion der Ziel-Nukleinsäuren, kombiniert mit einem innovativen Sensormodul. Dabei werden die einzelnen Basen zu Surrogatpolymeren, sogenannten Xpandomeren, ausgedehnt. Diese Reportermoleküle sind vielfach länger als die ursprüngliche Nukleinsäuresequenz und erzeugen daher in dem speziellen Nanopore-Sequenzierungsverfahren besonders starke Signale mit minimalem Hintergrundrauschen. Laut Roche ermöglicht der SBX-Ansatz eine sehr schnelle, genaue und flexible Bestimmung der DNA-Sequenz.
„Die Wissenschaft hinter der SBX-Technologie stellt einen bedeutenden Durchbruch dar, der die Einschränkungen bestehender Sequenzierungslösungen überwindet“, erklärt Matt Sause, CEO von Roche Diagnostics. „Durch die Integration und Weiterentwicklung zweier Technologien hat Roche mit SBX einen differenzierten Ansatz geschaffen, der unvergleichliche Geschwindigkeit, Effizienz und Flexibilität bietet. Die Geschwindigkeit und Genauigkeit von SBX hat das Potential, die Anwendung der Sequenzierung in Forschung und Gesundheitswesen zu revolutionieren.“
Die Technologie soll für eine Vielzahl von Anwendungen in der Genom-, Exom- und RNA-Sequenzierung einsetzbar sein. Bis zu sieben vollständige Genome pro Stunde bei 30-facher Abdeckung sollen damit sequenziert werden können, was fünf Milliarden Duplex-Reads pro Stunde – in beiden zugleich sequenzierten DNA-Strängen – entspricht.
Zwei entscheidende Neuentwicklungen
Die erste Innovation ist der Expansionsansatz von SBX, entwickelt von Mark Kokoris und Robert McRuer beim US-Biotech-Unternehmen Stratos Genomics, das Roche 2020 übernommen hatte. Das Ziel war, die Limitierung herkömmlicher Methoden zu überwinden, die in der dichten Packung der Nukleinsäure-Sequenzen liegt und in schlechten Signal-zu-Rausch-Verhältnissen resultiert. „Die Lösung der Signal-Rausch-Problemstellung ist ein entscheidender Effizienzfaktor für diese Technologie. Dank dieser Fähigkeit können wir flexibel über verschiedene Durchsatzniveaus hinweg mit demselben Sequenzierungssystem arbeiten, was den Nutzern einen erheblichen Vorteil bietet“, so Kokoris, Leiter der SBX-Technologie bei Roche.
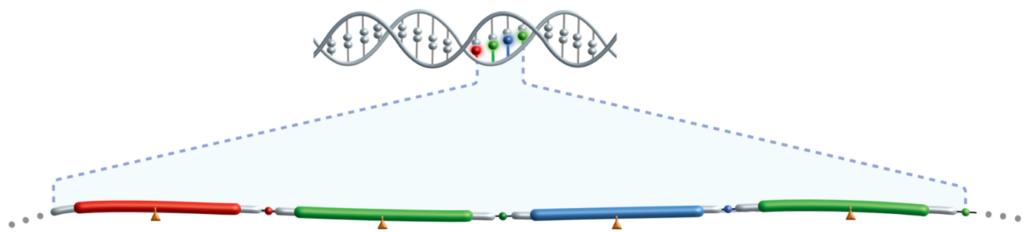
Hierfür werden die Sequenzen für die anschließende Nanoporen-Sequenzierung zunächst zu Xpandomeren erweitert. Die Basis stellen expandierbare Nukleosidtrisphosphate dar, die X-NTPs, welche signalstarke Reporter-Codes enthalten. Eine speziell entwickelte Polymerase integriert die X-NTP-Moleküle in das wachsende Xpandomer – stabilisiert durch Polymerase-Enhancer (PEMs). Nach der Synthese werden die Bindungen im Xpandomer gespalten und ein langes Molekül ausgestreckt, das etwa fünfzigmal länger ist als die ursprüngliche Sequenz. Somit werden die Einzelstränge in der Reaktion ergänzt, im Gegensatz zur PCR allerdings nicht amplifiziert. Wenngleich auch diese Methode nicht völlig fehlerfrei ist, gibt Roche für Einzelnukleotid-Varianten eine Sequenziergenauigkeit von mindestens 99,8% an.
Die andere Innovation betriff das Auslesen der Sequenzen. Nach der Synthese wird das Xpandomer über eine Nanopore geleitet, wobei Spannungspulse seine Bewegung steuern und die Reporter-Codes mit hoher Präzision ausgelesen werden. Dies geschieht über ein von Genia Technologies entwickeltes Hochdurchsatz-Sensormodul, das auf der „Complementary Metal Oxide Semiconductor“ (CMOS)-Technologie beruht und eine ultraschnelle und parallele Verarbeitung ermöglichen soll.

 Roche
Roche Roche
Roche Jan Krönke
Jan Krönke